Académique Documents
Professionnel Documents
Culture Documents
Activité 11 - ARN, Molécule Intermédiaire
Transféré par
ykwt7nhrhhTitre original
Copyright
Formats disponibles
Partager ce document
Partager ou intégrer le document
Avez-vous trouvé ce document utile ?
Ce contenu est-il inapproprié ?
Signaler ce documentDroits d'auteur :
Formats disponibles
Activité 11 - ARN, Molécule Intermédiaire
Transféré par
ykwt7nhrhhDroits d'auteur :
Formats disponibles
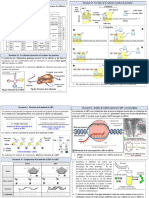
NOM : Activité 11 : L’ARN, molécule messagère
Capacités : Etape A Etape B
Comprendre des expériences historiques
Utiliser des logiciels de données moléculaires
L’expression d’un gène conduit à la synthèse d’une protéine. Celle-ci se déroule 3 étapes :
La première étape correspond à la formation d’une molécule intermédiaire, l’ARNm, nécessaire à la synthèse des
protéines. On veut montrer le rôle clé de cette molécule au cours de la synthèse des protéines.
Expériences préliminaires page 106-107 :
A partir des résultats des expériences décrites à la page 106-107, valider ou corriger les affirmations suivantes :
Doc a : D’après cette expérience, on peut dire que :
1. Les points noirs correspondent à l’incorporation d’acides aminés radioactifs dans les protéines.
2. La synthèse des protéines se fait dans le noyau.
Doc b : La coloration au vert de méthyle-pyronine de la cellule d’oignon permet :
1. De mettre en évidence que l’ARN est présent dans le cytoplasme.
2. De dire que l’ADN est passé dans le cytoplasme.
Doc c : les expériences de Jacob et Monod confirment que
1. L’addition de lactose dans le milieu induit la synthèse de la protéine qui s’effectue avant la synthèse
d’ARNm.
2. L’injection d’ARNm, déclenche la synthèse de la protéine dans un milieu sans lactose.
Doc 2 page 107 : Décrire les résultats de cette expérience.
Etape A : Suivre le protocole :
1. Pour comparer les molécules d’ADN et ARN.
Matériel : logiciel Libmol ; ouvrir deux onglets et les fichiers ADN et ARNm.
ADN ARNm
Bases azotées Adénine, thymine, guanine, cytosine
Sucre Désoxyribose
Acide phosphorique Présent
Représentation d’un nucléotide P-D-A ou P-D-T ou P-D-G ou P-P-C
Structure générale 2 chaines de nucléotides complémentaires
Nom complet de la molécule Acide désoxyribonucléique
2. Pour décrire la synthèse d’ARNm appelée transcription.
Elle s’effectue à partir d’un des deux brins d’ADN (brin transcrit) grâce à une enzyme ARN polymérase.
a) Charger fichier ARN polymérase en cours de transcription dans Libmol ;
b) Afficher en ruban ;
c) Eteindre la protéine ;
d) Colorer par résidus
On peut en déduire que l’ARN formé est une séquence :
De nucléotides d’ARN complémentaires de la séquence du brin transcrit d’ADN
De nucléotides d’ARN identiques à la séquence du brin transcrit d’ADN.
Appeler le professeur
3. Pour comparer l’ARN pm (sortant de transcription) et l’ARNm présent dans le cytoplasme
Matériel : logiciel Génigen :
a) Charger le fichier « maturation de l’ARN, chaîne de l’hémoglobine
b) Comparer les séquences
c) Proposer une hypothèse pour expliquer les différences constatées.
4. Pour établir une correspondance entre l’ARNm et la protéine
Matériel : logiciel Génigen :
a) Charger les molécules : Protéine, chaine béta avant modification post transductionnelle et ARNm de la chaine
béta de l’hémoglobine
b) Sélectionner la séquence d’ARN et la traduire en séquence d’acides aminés (aller dans action) ; Décrire le
résultat
c) Choisir la relation qui semble la plus probable entre la séquence de nucléotides d’ARN et la séquence d’acides
aminés en justifiant :
à 1 nucléotide correspond un acide aminé
à 2 nucléotides consécutifs correspond un acide aminé
à 3 nucléotides consécutifs correspond un acide aminé
d) Vérifier cette hypothèse en affichant le code génétique et en identifiant le ou les codon (s) correspondant à :
Met :
Glu :
Val :
e) Identifier la particularité des codons UAA ou UAG ou UGA du code génétique :
Dans fichier, saisir une séquence d’ARNm de 30 nucléotides de votre choix, en mettant en position 16, 17, 18, les
codons UAA ou UAG ou UGA puis sélectionner la séquence et la traduire.
Séquence ARNm :
Séquence d’acides aminés :
Identifier la particularité des codons UAA ou UAG ou UGA :
Bilan : Compléter le schéma fonctionnel avec les étapes qui conduisent à partir de d’une séquence d’ADN, à la sortie
d’une molécule d’ARNm traduite en protéine
Vous aimerez peut-être aussi
- Ciam 1SMDocument13 pagesCiam 1SMaboubacarsilecamara92Pas encore d'évaluation
- Série d'exercices N°1 - SVT expression de l'I G, genigénétique et mutation - 3ème Sciences exp (2023-2024) Mr مصباح العربيDocument4 pagesSérie d'exercices N°1 - SVT expression de l'I G, genigénétique et mutation - 3ème Sciences exp (2023-2024) Mr مصباح العربيkadra55amri100% (1)
- Chapitre 2 Nature Et Expression de L-Information G-N-TiqueDocument33 pagesChapitre 2 Nature Et Expression de L-Information G-N-Tiqueal mounir alaouiPas encore d'évaluation
- Mécanisme de L'expression de L'information Génétique Transcription Et TraductionDocument6 pagesMécanisme de L'expression de L'information Génétique Transcription Et Traductionsquiggle2238Pas encore d'évaluation
- Série Génétique - 1bac SMDocument4 pagesSérie Génétique - 1bac SMouiamePas encore d'évaluation
- BG3 ExosDocument22 pagesBG3 Exospersonne12Pas encore d'évaluation
- SVT 1ère C - L6 - La Synthèse Des ProtéinesDocument16 pagesSVT 1ère C - L6 - La Synthèse Des ProtéinesNamoryPas encore d'évaluation
- Ciam 1SMDocument10 pagesCiam 1SMaboubacarsilecamara92Pas encore d'évaluation
- Synthese Des ProteinesDocument2 pagesSynthese Des Proteinesdiawbachir15Pas encore d'évaluation
- Unité 2 Chapitre 2 Docs 2Document4 pagesUnité 2 Chapitre 2 Docs 2maeitfatimaPas encore d'évaluation
- Controle 3 de SVT 1er TrimestreDocument2 pagesControle 3 de SVT 1er Trimestreelfiguigui2011100% (1)
- SynthèseDocument10 pagesSynthèseElvy prices Sampinou mpompaPas encore d'évaluation
- La Synthèse ProtéiqueDocument6 pagesLa Synthèse ProtéiquealiPas encore d'évaluation
- Cours Th1 Chap 5 L'expression Du Patrimoine GénétiqueDocument24 pagesCours Th1 Chap 5 L'expression Du Patrimoine Génétique5cyfwcw8tzPas encore d'évaluation
- Objectif Terminal: Expliquer Les Différents Mécanismes Intervenants Dans La Synthèse Des ProtéinesDocument2 pagesObjectif Terminal: Expliquer Les Différents Mécanismes Intervenants Dans La Synthèse Des ProtéinesBenji JeanPas encore d'évaluation
- Exe171 SynthDocument6 pagesExe171 SynthMahmoud WattPas encore d'évaluation
- Exe171 SynthDocument6 pagesExe171 SynthModou LôPas encore d'évaluation
- Capture D'écran . 2023-04-27 À 15.59.03Document4 pagesCapture D'écran . 2023-04-27 À 15.59.03tehillahngolo3Pas encore d'évaluation
- CM04 - Traduction Et Routage Des ProteinesDocument8 pagesCM04 - Traduction Et Routage Des ProteinesRock In BandPas encore d'évaluation
- 4expression de L Information Genetique Activites 1Document5 pages4expression de L Information Genetique Activites 1KoffiPas encore d'évaluation
- Activité 7 Traduction Chapitre 1 Unité 2Document3 pagesActivité 7 Traduction Chapitre 1 Unité 2hamamielloPas encore d'évaluation
- Séance D'exercice 2022Document5 pagesSéance D'exercice 2022Abdou azizouPas encore d'évaluation
- Devoir de Contrôle N°1 Avec Correction - SVT - 3ème Sciences Exp (2023-2024) MR HAOUARI ALIDocument6 pagesDevoir de Contrôle N°1 Avec Correction - SVT - 3ème Sciences Exp (2023-2024) MR HAOUARI ALInediasakliPas encore d'évaluation
- Controle 2pc Unite 2Document4 pagesControle 2pc Unite 2Imane S.100% (1)
- Cellules Chrxgenes Partie 3 PDFDocument45 pagesCellules Chrxgenes Partie 3 PDFMohamed MarsoulPas encore d'évaluation
- Base de L'hereditéDocument67 pagesBase de L'hereditéNanitta ChhadePas encore d'évaluation
- 3 Serie Td3 La Synthese Des Proteines 2016 2017Document2 pages3 Serie Td3 La Synthese Des Proteines 2016 2017Houleye Kane100% (4)
- 3 Serie td3 La Synthese Des Proteines 2016 2017Document2 pages3 Serie td3 La Synthese Des Proteines 2016 2017Houleye KanePas encore d'évaluation
- UE1 - Biochimie - Expression Du GénomeDocument45 pagesUE1 - Biochimie - Expression Du Génomesun-nee-chan9Pas encore d'évaluation
- Cours 2 MTDocument65 pagesCours 2 MTحسن غالية حسن غاليةPas encore d'évaluation
- TD RecapitulatifDocument4 pagesTD RecapitulatifZa Hra100% (2)
- Biologie MoléculaireDocument10 pagesBiologie Moléculairemalak wxPas encore d'évaluation
- Cours Transcription S5Document46 pagesCours Transcription S5ونزار عبد القادرPas encore d'évaluation
- IV. La Traduction: Document 1: Les Différents Acteurs Intervenant Dans La Synthèse D'une Protéine: La TRADUCTIONDocument4 pagesIV. La Traduction: Document 1: Les Différents Acteurs Intervenant Dans La Synthèse D'une Protéine: La TRADUCTIONFadhel SlamaPas encore d'évaluation
- Tweet: La Synthèse Des ProtéinesDocument59 pagesTweet: La Synthèse Des ProtéinesNadaPas encore d'évaluation
- SVT 1ère D - L5 - La Synthèse Des ProtéinesDocument12 pagesSVT 1ère D - L5 - La Synthèse Des ProtéinesYedson GrâcePas encore d'évaluation
- Chapitre 03Document5 pagesChapitre 03You NesPas encore d'évaluation
- J3 - 7. Outils Et Technique de La BiomoleDocument80 pagesJ3 - 7. Outils Et Technique de La Biomolezz9jm8kmp7Pas encore d'évaluation
- Biologie Moléculaire Et GG 2Document30 pagesBiologie Moléculaire Et GG 2Sangaré soungaloPas encore d'évaluation
- La Transcription l3 Microbiologie 2022Document8 pagesLa Transcription l3 Microbiologie 2022rou röuPas encore d'évaluation
- A Synthèse Des ProtéinesDocument3 pagesA Synthèse Des ProtéinesNadaPas encore d'évaluation
- Les Ribosomes Et La Synthèse Protéique B6Document10 pagesLes Ribosomes Et La Synthèse Protéique B6kim namjoonPas encore d'évaluation
- Le Code Génétique: Un Système Universel de Correspondance Entre ADN Et Protéine. (Pages 108/109)Document4 pagesLe Code Génétique: Un Système Universel de Correspondance Entre ADN Et Protéine. (Pages 108/109)MuhamedMouslimPas encore d'évaluation
- BioMol Med2 Chap1-14 05 2022Document54 pagesBioMol Med2 Chap1-14 05 2022Denisco NkemgnePas encore d'évaluation
- Diaporama Expression GénétiqueDocument26 pagesDiaporama Expression GénétiqueLyna rbhPas encore d'évaluation
- Reproduction Du Génotype Au PhénotypeDocument7 pagesReproduction Du Génotype Au Phénotypeorion.13ferrPas encore d'évaluation
- ED 2 de Biologie Cellulaire FMMDocument5 pagesED 2 de Biologie Cellulaire FMMbmnkhalilPas encore d'évaluation
- EXEMPLE DE DEVOIR TYPE BAC PREMIERE S SVT - THEMES 1 Et 2Document3 pagesEXEMPLE DE DEVOIR TYPE BAC PREMIERE S SVT - THEMES 1 Et 2Houleye KanePas encore d'évaluation
- L Expression Du Patrimoine Genetique - Fiche de CoursDocument11 pagesL Expression Du Patrimoine Genetique - Fiche de CoursmillotPas encore d'évaluation
- SVT TA - L7 - La Biosynthèse Des ProtéinesDocument12 pagesSVT TA - L7 - La Biosynthèse Des Protéinestangaramariam566Pas encore d'évaluation
- Exercices Sur L'information GénétiqueDocument4 pagesExercices Sur L'information Génétiquedraboissouf174Pas encore d'évaluation
- Replication - A La ProteineDocument18 pagesReplication - A La ProteineAmenjulio YovoPas encore d'évaluation
- Synthèses Des ProtéinesDocument1 pageSynthèses Des ProtéinesAl sarahPas encore d'évaluation
- TP N°5 (1ère S)Document2 pagesTP N°5 (1ère S)sandra.le.du100% (2)
- EXPRESSION DE L'INFORMATION GÉNÉTIQUE TD (Activités)Document28 pagesEXPRESSION DE L'INFORMATION GÉNÉTIQUE TD (Activités)kamel Daouthi100% (1)
- Début Chapitre 3 Expression Du Patrimoine Génétique 2023-2024Document3 pagesDébut Chapitre 3 Expression Du Patrimoine Génétique 2023-2024cloe.garrotPas encore d'évaluation
- Fusionné 20240306 1719Document11 pagesFusionné 20240306 1719Mouhamed lamine SonkoPas encore d'évaluation
- Acides nucléiques: Les Grands Articles d'UniversalisD'EverandAcides nucléiques: Les Grands Articles d'UniversalisPas encore d'évaluation
- Au-delà de l'ADN : La Révolution Épigénétique: Des Mécanismes Cellulaires aux Facteurs Environnementaux : Comment l'Épigénétique Façonne Notre Destin Biologique et les Implications pour la Santé, le Comportement et l'Avenir de la RechercheD'EverandAu-delà de l'ADN : La Révolution Épigénétique: Des Mécanismes Cellulaires aux Facteurs Environnementaux : Comment l'Épigénétique Façonne Notre Destin Biologique et les Implications pour la Santé, le Comportement et l'Avenir de la RecherchePas encore d'évaluation
- BlésDocument7 pagesBlésoksaidrachid989Pas encore d'évaluation
- Extraction Des Huiles Essentielles D'une Substance NaturelleDocument7 pagesExtraction Des Huiles Essentielles D'une Substance NaturelleSmail ToumiPas encore d'évaluation
- Document??Document3 pagesDocument??Fayssal MerainiPas encore d'évaluation
- Tse ChimieDocument67 pagesTse Chimiefombafousseini46Pas encore d'évaluation
- Jean Louis CorrigéDocument40 pagesJean Louis CorrigéEspoirPas encore d'évaluation
- Mimoire Safran CsDocument55 pagesMimoire Safran CsAbdelghafour FarisPas encore d'évaluation
- CHROMATOGRAPHIE LIQUIDE (Pénétration Du Gel)Document25 pagesCHROMATOGRAPHIE LIQUIDE (Pénétration Du Gel)Oussama El BouadiPas encore d'évaluation
- 11 Les Vegetaux FraisDocument15 pages11 Les Vegetaux Fraisjean jacques StéphaniePas encore d'évaluation
- DefinitionDocument8 pagesDefinitionmeriemPas encore d'évaluation
- TP MiniprepDocument6 pagesTP MiniprepBOURRAKH HAMZAPas encore d'évaluation
- Covasilic 15 TdsDocument1 pageCovasilic 15 TdsSotiris SpanopoulosPas encore d'évaluation
- Chapitre 4Document18 pagesChapitre 4zohra yellasPas encore d'évaluation
- Corrigés Leçon 6 - Composés Oreganiques OxygénésDocument5 pagesCorrigés Leçon 6 - Composés Oreganiques Oxygénésholyeric50Pas encore d'évaluation
- Rapport PS RUNI Rapport PS - EvDocument19 pagesRapport PS RUNI Rapport PS - EvKhalid MargaaPas encore d'évaluation
- Memoire FinalDocument53 pagesMemoire FinalCandice REYPas encore d'évaluation
- Aurore 1ere C&DDocument280 pagesAurore 1ere C&DPascaline Magnan90% (10)
- La PollutionDocument20 pagesLa PollutionSamõ BibäPas encore d'évaluation
- Catalogue 2017 Trionyx - Universal Technique 2018Document136 pagesCatalogue 2017 Trionyx - Universal Technique 2018UNIVERSAL TECHNIQUEPas encore d'évaluation
- EXERCICES BiochimieDocument15 pagesEXERCICES Biochimiesaiddbilij6Pas encore d'évaluation
- TP de Chimie Organique FinDocument10 pagesTP de Chimie Organique FinEliel Newton100% (1)
- Mémoire Magister MessaoudiDocument82 pagesMémoire Magister MessaoudiAhmed CharPas encore d'évaluation
- Santé Et EnvironnementDocument11 pagesSanté Et Environnementohtmane3Pas encore d'évaluation
- Legenda Sedie ChairsDocument18 pagesLegenda Sedie ChairsJaymon P KPas encore d'évaluation
- PET Semi CristallinDocument3 pagesPET Semi CristallinNicolas TETARD100% (1)
- Tga 2015 Pour SiteDocument4 pagesTga 2015 Pour SiteOthman ZwPas encore d'évaluation
- Rapport de Stage COGB (Enregistré Automatiquement) 1Document23 pagesRapport de Stage COGB (Enregistré Automatiquement) 1serine serine100% (2)
- Différents Types de Vieillissement Chimique Des PlastiquesDocument14 pagesDifférents Types de Vieillissement Chimique Des PlastiquesFadila Amara100% (2)
- Lab Gta 26Document24 pagesLab Gta 26Neo MaximusPas encore d'évaluation
- TP CosmetoDocument12 pagesTP CosmetoKlinnanTourePas encore d'évaluation
- Traitement Et EpurDocument88 pagesTraitement Et EpurDadi BrizPas encore d'évaluation