Académique Documents
Professionnel Documents
Culture Documents
Méthodes de Détection Et D'identification Des HPV
Transféré par
Mezouar AbdennacerTitre original
Copyright
Formats disponibles
Partager ce document
Partager ou intégrer le document
Avez-vous trouvé ce document utile ?
Ce contenu est-il inapproprié ?
Signaler ce documentDroits d'auteur :
Formats disponibles
Méthodes de Détection Et D'identification Des HPV
Transféré par
Mezouar AbdennacerDroits d'auteur :
Formats disponibles
PAPILLOMAVIRUS
THÉMATIQUE À TAPER
(HPV)
Méthodes de détection et d’identification
des HPV
Laurence Olliera, Valérie Giordanengoa,b,*
RÉSUMÉ
SUMMARY
P
Parmi i lles papillomavirus
ill i h
humains
i (HPV)
(HPV), on di
distingue
ti lles HPV dit
dits à h
hautt
risque oncogénique et les HPV à bas risque oncogénique. L’association Detection and identification of HPV
de ces génotypes à haut risque oncogénique et du cancer du col de l’uté- Among human papillomaviruses (HPV), high risk
rus est aujourd’hui bien établie et permet de proposer des techniques de (HR) and low risk HPV types can be distinguished.
détection de ces génotypes d’HPV. Ce diagnostic d’infection est réalisé The well established association of HR-HPV with
par des techniques de biologie moléculaire qui reposent sur le principe cervical cancer justifies the detection and identifi-
de l’hybridation moléculaire entre : un ADN ou ARN cible et une sonde cation of these HR-HPV genotypes. This diagno-
spécifique. Elles permettent de détecter, quantifier ou génotyper le ou les sis of infection is realized by molecular biology
HPV présents. tolls based on molecular hybridization between
DNA or RNA target and specific probes. They
HPV – détection – génotypage – quantification – PCR. allow detection, quantification or typing of one
or several HPV genotypes.
HPV – detection – genotyping – quantification – PCR.
1. Introduction
Les papillomavirus humains (HPV) sont des virus à ADN
infectant les kératinocytes des muqueuses épidermoïdes
génitales et de la peau [16, 20, 21]. Parmi les génotypes
d’HPV (près de 120), une trentaine environ présente un virale. Ces modèles ne peuvent pas être utilisés en routine
tropisme génital. Parmi eux, on distingue d’une part les pour le diagnostic et seules des techniques de biologie
HPV dits à haut risque oncogénique (high-risk HPV, HR- moléculaire permettent d’identifier qualitativement ou de
HPV), représentés par HPV16, 18, 31, 33, 35, 39, 45, 51, quantifier les séquences nucléiques des HPV.
52, 56, 58,59, 68, 73 et 82, et d’autre part, les HPV à bas Ces techniques de biologie moléculaire sont nombreuses
risque (low-risk HPV, LR-HPV) représentés par HPV 6, 11, et parfois complémentaires [19, 28] ; elles reposent sur le
40, 42, 43, 44, 54, 61, 70, 72, 81 et 89 [21, 22]. Le cycle principe de l’hybridation moléculaire entre un ADN ou un
viral des papillomavirus dépend de la différenciation épithé- ARN messager (ARNm) cible et une sonde spécifique.
liale et les méthodes d’isolement viral par culture cellulaire Certaines de ces techniques sont désormais utilisables
sont difficiles à mettre en place. Néanmoins, l’utilisation en routine de laboratoire. L’ADN génomique des HPV est
de modèles de culture organotypique de kératinocytes la cible la plus utilisée, car les ARNm sont plus fragiles.
ou de lambeaux tissulaires sur un gel de collagène ont Les étapes d’extraction et de dénaturation de l’ADN ou
permis des avancées significatives dans la compréhen- de l’ARNm cible sont éventuellement suivies d’une étape
sion des mécanismes moléculaires régulant la réplication d’amplification, précédant l’hybridation moléculaire. L’étape
ultérieure de détection des hybrides « cible-sonde » permet
de révéler la présence de la cible. Ainsi schématiquement,
les techniques de biologie moléculaire actuellement appli-
cables en routine au laboratoire sont réalisées en 3 ou
a Laboratoire de virologie 4 temps (extraction, +/- amplification, hybridation, révé-
Hôpital de l’Archet II lation), à partir d’un prélèvement effectué au niveau de la
151, route de Saint-Antoine-de-Ginestière – B.P. 3079 zone de jonction endocol-exocol (figure 1).
06202 Nice cedex 3
b Inserm U895 Biologie des interactions hôte-microorganisme – C3M
151, route Saint Antoine-de-Ginestière – B.P. 2 3094
2. Détection qualitative
06204 Nice cedex 3 du génome HPV
* Correspondance Lors de la détection qualitative des HPV, deux types de
giordane@unice.fr résultats peuvent être obtenus : soit le résultat indique
la présence (ou l’absence) de HR-HPV sans préciser le
article reçu le 13 mars, accepté le 20 mars 2008. génotype exact, soit le résultat précise le génotype de
© 2008 – Elsevier Masson SAS – Tous droits réservés. l’HPV détecté.
REVUE FRANCOPHONE DES LABORATOIRES - SEPTEMBRE/OCTOBRE 2008 - N°405 // 51
2.1. Détection des HPV à haut risque, captés sur la paroi de la microplaque. Après capture des
sans précision du type hybrides, un anticorps anti-duplex couplé à une phospha-
Ces techniques permettent de détecter la présence d’un tase alcaline (PAL) réagit avec la partie libre des duplex
ou de plusieurs HPV sans identifier le ou les génotypes (figure 2). En présence d’un substrat chimioluminescent,
précis présents dans l’échantillon. la PAL déclenche une émission de lumière détectée par
un luminomètre [2]. La sensibilité de cette technique est
2.1.1. Hybridation en phase liquide proche de celle de la PCR (polymerase chain reaction) [3,
La spécificité diagnostique de cette technique ancienne 26] tout en étant une technique rapide, reproductible et
repose sur une étape d’hybridation en solution, c’est applicable à de grandes séries.
une immunocapture en phase liquide. L’ADN est extrait Le rendu du résultat est qualitatif :
et l’hybridation en phase liquide est réalisée entre l’ADN - test HR-HPV positif : présence d’HPV à haut risque onco-
viral cible dénaturé et un « cocktail » de sondes ARN com- génique,
plémentaires de 13 HR-HPV (HPV16, 18, 31, 33, 35, 39, - test HR-HPV négatif : absence d’HPV à haut risque onco-
45, 51, 52, 56, 58,59, 68). Les hybrides ADN/ARN formés génique.
sont stables et, grâce à des anticorps polyclonaux anti-
duplex ADN/ARN fixés sur la paroi des puits, ils sont 2.1.2. Amplification par PCR couplée à une détection
immunoenzymatique des amplicons
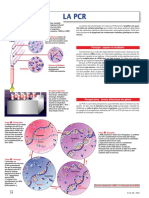
Figure 1 – Représentation schématique des différentes étapes De nombreuses stratégies existent, toutes utilisent une étape
des techniques de détection et de génotypage des HPV. d’amplification d’une séquence caractéristique du génome
HPV, puis une étape de révélation de ces amplicons grâce
à une réaction colorimétrique [4, 23]. Ainsi sont successi-
vement réalisées : la lyse et l’extraction de l’ADN contenu
dans les cellules cervicales, puis la PCR avec des amorces
biotinylées encadrant un fragment de la région L1. Le choix
de cette région L1 permet une PCR de genre amplifiant le
fragment ADN quel que soit le génotype d’HPV. L’amplicon
est ensuite dénaturé puis hybridé, dans un puits, avec un
cocktail de sondes spécifiques et complémentaires des
13 HR-HPV. Les hybrides capturés dans les puits sont révélés
par réaction colorimétrique grâce au groupement biotine
présent sur l’amplicon (figure 3). Le rendu du résultat est
qualitatif : présence, ou non, d’HPV à haut risque oncogéni-
que dans le prélèvement. Dans la plupart des cas, une PCR
contrôle qui amplifie une région d’un ADN cellulaire ubiqui-
taire est réalisée afin de valider la qualité du prélèvement et
les conditions d’amplification et de révélation.
2.2. Génotypage des HR-HPV
Réalisées en 3 temps (extraction, hybridation, révélation) ou 4 temps (extraction, Le patient pouvant être infecté par un seul type d’HPV ou
amplification, hybridation, révélation).
présenter une infection multiple, ces techniques identifient
Figure 2 – Hybridation en phase liquide. Figure 3 – Détection immunoenzymatique
des amplicons HPV .
L’ADN extrait dénaturé est hybridé avec les sondes ARN, puis capturé sur le puits L’ADN extrait dénaturé est capturé sur le puits par des sondes
par des anticorps anti-hybride ADN/ARN et révélé. oligonucléotidiques puis révélé.
52 // REVUE FRANCOPHONE DES LABORATOIRES - SEPTEMBRE/OCTOBRE 2008 - N°405
PAPILLOMAVIRUS (HPV)
le (ou les) génotype(s) d’HPV présents dans l’échantillon profils de tous les génotypes d’HPV. La localisation de la
analysé. Elles comportent une étape d’amplification PCR bande (ou des bandes) colorée(s) sur la bandelette permet
d’un fragment d’ADN HPV. Le choix de la région amplifiée de définir le (ou les) génotype(s) présent(s) dans le prélève-
doit répondre à deux contraintes [5, 11, 25] : une séquence ment [10, 27]. Ces techniques portent le nom de line blot
ADN suffisamment conservée à ses extrémités afin de per- assay [10], line probe assay (LiPA) [27] ou de linear array
mettre une PCR de genre, mais divergente dans sa région assay [29] La sensibilité de ces tests permet la détection
interne pour autoriser le génotypage par comparaison avec simultanée de plusieurs génotypes d’HPV [6, 29].
des séquences de génotypes connus.
La réalisation d’une PCR contrôle, à partir d’un ADN cel- 2.2.3. Génotypage par puce à ADN
lulaire ubiquitaire, peut valider la qualité du prélèvement Cette technique de biologie moléculaire est en plein essor
et le bon déroulement des étapes d’amplification et de et permet un génotypage rapide [1, 12, 14]. La PCR HPV de
révélation. genre est réalisée avec des amorces fluorescentes, puis le
produit PCR est hybridé sur une lame. Cette lame contient
2.2.1. Génotypage par séquençage les sondes spécifiques des génotypes d’HPV recherchés
C’est la méthode de référence puisque c’est la séquence déposées sous forme de spots bien distincts. Les puces
complète de la région L1 qui définit les différents génoty- applicables au diagnostic sont des puces de basse densité,
pes d’HPV. Cependant, en routine, le génotype est réalisé avec 5 à 100 sondes différentes par lame. Le produit PCR
à partir d’une plus courte séquence, celle des amplicons : étant fluorescent aucune étape de révélation n’est nécessaire
région variable suivant le type qui sera alignée avec des et, après lavage de la lame, la lecture se fait directement sur
séquences de référence des différents génotypes d’HPV un scanner qui localise l’hybride cible-sonde et en déduit le
[7, 15]. génotype (figure 5). Cette technique est simple, sensible
Cette méthode permet de détecter le génotype d’HPV et rapide. Elle permet également de détecter les différents
majoritaire, présent dans le prélèvement, mais peut diffici- types dans les infections HPV multiples.
lement diagnostiquer les infections multiples. En revanche,
elle permet de détecter des variants d’HPV (pour les HPV 2.2.4. Génotypage par la technologie Luminex®
16 et 18 principalement) dont la distribution varie selon La technologie Luminex® est une nouvelle technologie de
les zones géographiques. cytométrie en flux permettant la détection de réactions
multiples dans un même tube. Ce système multi-analyti-
2.2.2. Génotypage par sondes immobilisées que est constitué d’un cytomètre de flux à 2 lasers et d’un
sur bandelettes ensemble de microsphères. Après amplification avec des
Dans cette technique, c’est lors de l’étape d’hybridation amorces fluorescentes (PCR de genre), l’ADN amplifié est
que l’on différencie les divers génotypes présents. La PCR hybridé sur les microsphères recouvertes chacune d’un
HPV est réalisée avec des amorces biotinylées. Les produits oligonucléotide spécifique d’un génotype d’HPV. Chaque
amplifiés sont hybridés sur une bandelette sur laquelle des type de microsphères est reconnaissable en cytométrie
oligonucléotides spécifiques de différents génotypes d’HPV en flux par un code-couleur spécifique et l’amplicon est
ont été préalablement fixés. Les hybrides amplicon - oligo- lui aussi marqué par un fluorophore. Il est donc possible,
nucléotide sont révélés par réaction colorimétrique grâce grâce à cette technologie, d’analyser simultanément jusqu’à
au groupement biotine présent sur l’amplicon (figure 4). 100 types de microsphères par condition et donc d’ana-
La lecture s’effectue ensuite, par comparaison entre le lyser rapidement les différents types d’HR-HPV présents
profil d’hybridation obtenu et une plaquette où figurent les dans le prélèvement [24].
Figure 4 – Génotypage par reverse dot-blot. Figure 5 – Génotypage par puce à ADN.
Les amplicons sont hybridés sur la bandelette formant des hybrides Les amplicons sont hybridés sur la lame formant des hybrides amplicon
amplicon – oligonucléotide spécifique d’un type d’HPV puis sont révélés – oligonucléotide spécifique d’un type d’HPV puis sont visualisés par le
par réaction colorimétrique. scanner à fluorescence.
REVUE FRANCOPHONE DES LABORATOIRES - SEPTEMBRE/OCTOBRE 2008 - N°405 // 53
3. Détection quantitative que, la détection des ARNm E6-E7 peut être un marqueur
intéressant pour apprécier le risque d’évolution vers une
du génome HPV lésion cancéreuse. Dans la technique NASBA (nucleic acid
sequence based amplification) [13, 18], les ARN viraux et
Ces techniques évaluent la charge virale d’un type précis cellulaires extraits des prélèvements cervicaux sont rétro-
d’HPV. La quantité de virus détecté dans les frottis pour- transcrits en ADN par la transcriptase inverse. L’une des
rait être liée au grade de la lésion et donc être un facteur deux amorces utilisées possède le promoteur de la T7-ARN
pronostique, mais les résultats sont parfois contradictoires polymérase et permet à cette T7-ARN polymérase de
[8, 9, 17]. L’arrivée de la PCR en temps réel permet de transcrire une centaine de copies d’ARN à partir d’une
nombreuses études. Cette technique donne une quantifi- copie d’ADN, réalisant une amplification isotherme des
cation précise (nombre de copies d’ADN) par comparaison ARNm. La détection de ces ARNm se fait par hybridation
avec des standards internes. La détection et la quantifi- avec des sondes oligonucléotidiques fluorescentes spé-
cation simultanées de l’ADN cible sont possibles grâce à cifiques de type, permettant le génotypage.
la mesure « en temps réel » de l’émission de fluorescence.
L’intensité de cette fluorescence est proportionnelle à la
quantité d’amplicons synthétisés et donc au nombre de
5. Conclusion
cibles (génome HPV) présentes initialement dans le pré-
lèvement. L’analyse de la pente de la courbe de mesure La détection des HPV à haut risque oncogénique est un
de la fluorescence, au tout début de la phase exponen- outil indispensable à la prise en charge des lésions du col
utérin. Dans les années à venir, les techniques de biologie
tielle, permet la quantification de cet ADN par rapport à
moléculaire serviront probablement dans le suivi de ces
une gamme étalon.
lésions du col utérin après traitement, et dans le diagnostic
d’autres lésions précancéreuses. L’évolution rapide des
4. Détection des ARNm HPV techniques nous permet actuellement d’avoir de multiples
moyens, parfois complémentaires, pour détecter, identifier
Puisque les protéines oncogéniques E6 et E7 sont res- et quantifier les HPV. Cependant les indications de ces
ponsables de la carcinogenèse liée aux HPV à haut ris- techniques doivent encore être évaluées.
[8] Fontaine J., Gravitt P., Duh L.M., Lefevre J., Pourreaux K., Hankins
Références C., Coutlee F., High level of correlation of human papillomavirus-16 DNA
viral load estimates generated by three real-time PCR assays applied
[1] Albrecht V., Chevallier A., Magnone V., Barbry P., Vandenbos F.,
on genital specimens, Cancer Epidemiol. Biomarkers Prev. 14 (2005)
Bongain A., Lefebvre J.C., Giordanengo V., Easy and fast detection 2200-2207.
and genotyping of high-risk human papillomavirus by dedicated DNA
[9] Gravitt P.E., Kovacic M.B., Herrero R., Schiffman M., Bratti C.,
microarrays, J. Virol. Methods 137 (2006) 236-244.
Hildesheim A., Morales J., Alfaro M., Sherman M.E., Wacholder
[2] Bergeron C., Jennel D., Poveda J., Cassonnet P., Orth G., Human S., Rodriguez A.C., Burk R.D., High load for most high risk human
papillomavirus testing in women with mild cytologic atypia, Obstet. papillomavirus genotypes is associated with prevalent cervical cancer
Gynecol. 95 (2000) 821-827. precursors but only HPV16 load predicts the development of incident
[3] Clavel C., Bory J.P., Rihet S., Masure M., Duval-Binninger I., Putaud disease, Int. J. Cancer 121 (2007) 2787-2793.
I., Lorenzato M., Quereux C., Birembaut, P., Comparative analysis of [10] Gravitt P.E., Peyton C.L., Apple R.J., Wheeler C.M., Genotyping of
human papillomavirus detection by hybrid capture assay and routine 27 human papillomavirus types by using L1 consensus PCR products
cytologic screening to detect high-grade cervical lesions, Int. J. Cancer by a single-hybridization, reverse line blot detection method, J. Clin.
75 (1998) 525-528. Microbiol. 36) (1998) 3020-3027.
[4] Clavel C., Rihet S., Masure M., Chypre C., Boulanger J.C., Quereux [11] Husnjak K., Grce M., Magdic L., Pavelic K., Comparison of five
C., Birembaut P., DNA-EIA to detect high and low risk HPV genotypes different polymerase chain reaction methods for detection of human
in cervical lesions with E6/E7 primer mediated multiplex PCR, J. Clin. papillomavirus in cervical cell specimens, J. Virol. Methods 88 (2000)
Pathol. 51 (1998) 38-43. 125-134.
[5] Coutlee F., Gravitt P., Kornegay J., Hankins C., Richardson H., [12] Hwang T.S., Jeong J.K., Park M., Han H.S., Choi H.K., Park T.S.,
Lapointe N., Voyer H., Franco E., Use of PGMY primers in L1 consensus Detection and typing of HPV genotypes in various cervical lesions by
PCR improves detection of human papillomavirus DNA in genital HPV oligonucleotide microarray, Gynecol. Oncol. 90 (2003) 51-56.
samples, J. Clin. Microbiol. 40 (2002) 902-907. [13] Kraus I., Molden T., Holm R., Lie A.K., Karlsen F., Kristensen G.B.,
[6] Didelot-Rousseau M.N., Courgnaud V., Nagot N., Ouedraogo A., Skomedal H., Presence of E6 and E7 mRNA from human papillomavirus
Konate I., Mayaud P., Weiss H., Van de Perre P., Segondy M., Comparison types 16, 18, 31, 33, and 45 in the majority of cervical carcinomas, J.
of INNO-LiPA HPV Genotyping V2 with PCR product subcloning and Clin. Microbiol. 44 (2006) 1310-1317.
sequencing for identification of genital human papillomavirus genotypes [14] Lee S.A., Kang D., Seo S.S., Jeong J.K., Yoo K.Y., Jeon Y.T., Kim J.W.,
in African women, J. Virol. Methods 135 (2006) 181-185. Park N.H., Kang S.B., Lee H.P., Song Y.S., Multiple HPV infection in cervical
[7] Feoli-Fonseca J.C., Oligny L.L., Filion M., Brochu P., Simard P., cancer screened by HPVDNAChip, Cancer Lett. 198 (2003) 187-192.
Russo P.A., Yotov W.V., A two-tier polymerase chain reaction direct [15] Lee S.H., Vigliotti V.S., Vigliotti, J.S., Pappu S., Routine human
sequencing method for detecting and typing human papillomaviruses in papillomavirus genotyping by DNA sequencing in community hospital
pathological specimens, Diagn. Mol. Pathol. 7(1998) 317-323. laboratories, Infect. Agent Cancer 2 (2007) 11.
54 // REVUE FRANCOPHONE DES LABORATOIRES - SEPTEMBRE/OCTOBRE 2008 - N°405
PAPILLOMAVIRUS (HPV)
[16] Longworth M.S., Laimins L.A., Pathogenesis of human detected by a non-radioactive general primer PCR mediated enzyme
papillomaviruses in differentiating epithelia, Microbiol. Mol. Biol. Rev. immunoassay, J. Clin. Pathol. 52 (1999) 17-22.
68(2004) 362-372. [24] Oh Y., Bae S.M., Kim Y.W., Choi H.S., Nam G.H., Han S.J.,
[17] Lorincz A.T., Castle P.E., Sherman M.E., Scott D.R., Glass A.G., ParkC.H., Cho Y., Han B.D., Ahn W.S., Polymerase chain reaction-
Wacholder S., Rush B.B., Gravitt P.E., Schussler J.E., Schiffman M., based fluorescent Luminex assay to detect the presence of human
Viral load of human papillomavirus and risk of CIN3 or cervical cancer, papillomavirus types, Cancer Sci. 98 (2007) 549-554.
Lancet 360. (2002) 228-229. [25] Qu W., Jiang G., Cruz Y., Chang C.J., Ho G.Y., Klein R.S., Burk
[18] Molden T., Kraus I., Skomedal H., Nordstrom T., Karlsen F., PreTect R.D., PCR detection of human papillomavirus: comparison between
HPV-Proofer: real-time detection and typing of E6/E7 mRNA from MY09/MY11 and GP5+/GP6+ primer systems, J. Clin. Microbiol. 35
carcinogenic human papillomaviruses, J. Virol. Methods 142) (2007) (1997) 1304-1310.
204-212. [26] Riethmuller D., Gay C., Bertrand X., Bettinger D., Schaal J.P.,
Carbillet J.P., Lassabe C., Arveux P., Seilles E., Mougin C., Genital human
[19] Molijn A., Kleter B., Quint W., van Doorn L.J., Molecular diagnosis
papillomavirus infection among women recruited for routine cervical
of human papillomavirus (HPV) infections, J. Clin. Virol. 32 Suppl 1
cancer screening or for colposcopy determined by Hybrid Capture II
(2005) S43-S51.
and polymerase chain reaction, Diagn Mol. Pathol. 8 (1999) 157-164.
[20] Munoz N., Human papillomavirus and cancer: the epidemiological
[27] van Doorn L.J., Quint W., Kleter B., Molijn A., Colau B., Martin M.T.,
evidence, J. Clin. Virol. 19 (2000) 1-5.
Kravang I., Torrez-Martinez N., Peyton C.L., Wheeler C.M. ,Genotyping
[21] Munoz N., Bosch F.X., de Sanjose S., Herrero R., Castellsague of human papillomavirus in liquid cytology cervical specimens by
X., Shah K.V., Snijders P.J., Meijer C.J., Epidemiologic classification of the PGMY line blot assay and the SPF(10) line probe assay, J. Clin.
human papillomavirus types associated with cervical cancer, N. Engl. J. Microbiol. 40 (2002) 979-983.
Med. 348 (2003) 518-527. [28] Vernon S.D., Unger E.R., Williams D., Comparison of human
[22] Munoz N., Castellsague X., de Gonzalez A.B., Gissmann L., Chapter papillomavirus detection and typing by cycle sequencing, line blotting,
1: HPV in the etiology of human cancer, Vaccine 24 Supp.3 (2006) S1- and hybrid capture, J. Clin. Microbiol. 38 (2000) 651-655.
S10. [29] Woo, Y.L., Damay, I., Stanley, M., Crawford, R., Sterling J., The
[23] Nindl I., Lotz B., Kuhne-Heid R., Endisch U., Schneider A., use of HPV linear array assay for multiple HPV typing on archival frozen
Distribution of 14 high risk HPV types in cervical intraepithelial neoplasia tissues and DNA specimens, J. Virol. Methods 142 (2007) 226-230.
REVUE FRANCOPHONE DES LABORATOIRES - SEPTEMBRE/OCTOBRE 2008 - N°405 // 55
Vous aimerez peut-être aussi
- SketchUp Pour Le Dessin Des MeublesDocument118 pagesSketchUp Pour Le Dessin Des MeublesSamantha Erickson100% (1)
- Reussir Vos Fraisages CNC3018 Pro CncFraises V1.0Document37 pagesReussir Vos Fraisages CNC3018 Pro CncFraises V1.0franck giorgi100% (1)
- Dossiers vaccination et politiques vaccinales: Les dossiers MorphéusD'EverandDossiers vaccination et politiques vaccinales: Les dossiers MorphéusPas encore d'évaluation
- Chapitre 4 Redressement Triphasé CommandéDocument11 pagesChapitre 4 Redressement Triphasé CommandéYahia Nino100% (1)
- Ob 2a860b Hs Trois Mariages Chez Les FortunesDocument215 pagesOb 2a860b Hs Trois Mariages Chez Les FortunesMentor MCconell RamsèsPas encore d'évaluation
- 5 - Traitement de L'hypertension ArtérielleDocument31 pages5 - Traitement de L'hypertension ArtérielleMezouar AbdennacerPas encore d'évaluation
- Carlos CASTANEDA - Histoires de PouvoirDocument188 pagesCarlos CASTANEDA - Histoires de Pouvoiralphaorio100% (1)
- Cours Pao 08.11Document30 pagesCours Pao 08.11tifenn.fleckPas encore d'évaluation
- Diagnostic. Virologique. LAFDocument52 pagesDiagnostic. Virologique. LAFmelchiPas encore d'évaluation
- Stratégies de Dépistage Des Lésions Précancéreuses Du Col de L'utérus Cytologie Ou Test HPVDocument9 pagesStratégies de Dépistage Des Lésions Précancéreuses Du Col de L'utérus Cytologie Ou Test HPVMezouar AbdennacerPas encore d'évaluation
- Classification Des Papillomavirus (HPV)Document3 pagesClassification Des Papillomavirus (HPV)Mezouar AbdennacerPas encore d'évaluation
- Methode de Diagnostic 10Document55 pagesMethode de Diagnostic 10MACON824100% (1)
- DiagbiolDocument14 pagesDiagbiolabdellah abdelkhalekPas encore d'évaluation
- Avant ProposDocument2 pagesAvant ProposMezouar AbdennacerPas encore d'évaluation
- Diagnostic Virus Respiratoires EcoleDocument64 pagesDiagnostic Virus Respiratoires EcoleessaidmaramnourPas encore d'évaluation
- Conférence de Dunarel BadescuDocument17 pagesConférence de Dunarel Badescuwilfried AGBETOPas encore d'évaluation
- HVP Et Cancer Classification Des LésionsDocument8 pagesHVP Et Cancer Classification Des LésionsMezouar AbdennacerPas encore d'évaluation
- 6 PapillomavirusDocument6 pages6 PapillomavirusOuattara BamoryPas encore d'évaluation
- BT1014 Ef1Document14 pagesBT1014 Ef1Deserted Shadow29Pas encore d'évaluation
- Biologie Moleculaire Dans La Prevention Du Risque Infectieux Chez Les Professionnels de Sante K ZeroualiDocument39 pagesBiologie Moleculaire Dans La Prevention Du Risque Infectieux Chez Les Professionnels de Sante K ZeroualiPekoJhames08gmail.comPas encore d'évaluation
- HPV PDFDocument3 pagesHPV PDFAnonymous VbEvDIOUaPas encore d'évaluation
- Vaccin Rna Anti Sars Cov2Document18 pagesVaccin Rna Anti Sars Cov2Mohamed Badreddine MokhtariPas encore d'évaluation
- P 32Document1 pageP 32greyPas encore d'évaluation
- 1 s2.0 S0929693X07787067 MainDocument6 pages1 s2.0 S0929693X07787067 MainCronos OttoPas encore d'évaluation
- HPV Et Cancers Mécanismes de L'oncogenèseDocument8 pagesHPV Et Cancers Mécanismes de L'oncogenèseMezouar AbdennacerPas encore d'évaluation
- Strategies Diag, Ostiques en VirologieDocument28 pagesStrategies Diag, Ostiques en VirologieTaoufik Ben HoumichPas encore d'évaluation
- Tratégie Diagnostique Des Hémopathies MalignesDocument6 pagesTratégie Diagnostique Des Hémopathies Malignesjallil jallilPas encore d'évaluation
- Bacterie MultiresistanteDocument11 pagesBacterie MultiresistanteNe ElPas encore d'évaluation
- Mtp-284450-Papillomavirus Les Virus Et La Physiopathologie de Linfection - Wo-dX38AAQEAAG4jpiEAAAAH-A.pdf Version 1uuuDocument15 pagesMtp-284450-Papillomavirus Les Virus Et La Physiopathologie de Linfection - Wo-dX38AAQEAAG4jpiEAAAAH-A.pdf Version 1uuuBektache nabylPas encore d'évaluation
- Immunité anti-HPV Et VaccinationDocument6 pagesImmunité anti-HPV Et VaccinationMezouar AbdennacerPas encore d'évaluation
- Tests de Diagnostic Rapides Des Hépatites Virales: Intérêts Et LimitesDocument5 pagesTests de Diagnostic Rapides Des Hépatites Virales: Intérêts Et LimitesHoussam DjeradPas encore d'évaluation
- Devoir BiotechnologieDocument8 pagesDevoir BiotechnologieRida MariyaPas encore d'évaluation
- Diagnostic VirologiqueDocument5 pagesDiagnostic VirologiqueLylia Bessire100% (2)
- VIRO 4 Diagnostic VirologiqueDocument16 pagesVIRO 4 Diagnostic VirologiquefsxnsstoaehkbhlzygPas encore d'évaluation
- M4-MB22. Virus À ARNDocument7 pagesM4-MB22. Virus À ARNسفير نعمونPas encore d'évaluation
- Cytomégalovirus Et EbvDocument29 pagesCytomégalovirus Et EbvSandrine WadePas encore d'évaluation
- Papillomavirus Humains (HPV) Et Cancers Associés Aspects ÉpidémiologiquesDocument8 pagesPapillomavirus Humains (HPV) Et Cancers Associés Aspects ÉpidémiologiquesMezouar AbdennacerPas encore d'évaluation
- Méthodes-De-Diagnostic-VirologiqueDocument4 pagesMéthodes-De-Diagnostic-VirologiquesarahPas encore d'évaluation
- 3 DG Infections ViralesDocument46 pages3 DG Infections ViralesAmenElleh ShilPas encore d'évaluation
- Evaluation Des Connaissances Des Médecins en Formation Au CHU Mohammed VI de Marrakech Vis A Vis Des Accidents Dexposition Au SangDocument1 pageEvaluation Des Connaissances Des Médecins en Formation Au CHU Mohammed VI de Marrakech Vis A Vis Des Accidents Dexposition Au Sanghiba samlaliPas encore d'évaluation
- Metaplasie CIN: Diu de ColposcopieDocument50 pagesMetaplasie CIN: Diu de ColposcopieMaria MogosPas encore d'évaluation
- Cours Herpes 2020-2021Document89 pagesCours Herpes 2020-2021Bouazzi IslemmPas encore d'évaluation
- Aqac126 303Document2 pagesAqac126 303Starix UkrPas encore d'évaluation
- Virus HerpesDocument8 pagesVirus HerpesKha LedPas encore d'évaluation
- 18-01-2023 Evaluation Clinique D'un Nouveau Test RT-PCR Pour LeDocument15 pages18-01-2023 Evaluation Clinique D'un Nouveau Test RT-PCR Pour Leprosper atebaPas encore d'évaluation
- Microbiologie Spéciale-Méd Dent-S9-Pr MAROUI 2023Document47 pagesMicrobiologie Spéciale-Méd Dent-S9-Pr MAROUI 2023Hamza BaibaiPas encore d'évaluation
- Présentation Raouiya Et Amina Pfe IA FinalDocument35 pagesPrésentation Raouiya Et Amina Pfe IA Finalsahlal aminaPas encore d'évaluation
- 18 Focus PCR Et ARN 16S Biomnis - 2Document2 pages18 Focus PCR Et ARN 16S Biomnis - 2adel strikePas encore d'évaluation
- Poster Pret Aporter 2.0Document1 pagePoster Pret Aporter 2.0Regoui ZinneddinePas encore d'évaluation
- 3 HerpesviridaeDocument22 pages3 HerpesviridaeRomi AtouiPas encore d'évaluation
- Exposé 4 GEDocument23 pagesExposé 4 GEFeriel AbzarPas encore d'évaluation
- BMGGDocument14 pagesBMGGMima UsmaPas encore d'évaluation
- CoursDocument7 pagesCourschamo chaimaPas encore d'évaluation
- 44505Document5 pages44505adelphe moukokoPas encore d'évaluation
- Avis SFM Valeur CT Excrétion Virale Version Finale 25092020Document10 pagesAvis SFM Valeur CT Excrétion Virale Version Finale 25092020Guillaume LavoixPas encore d'évaluation
- Memoire FcuDocument108 pagesMemoire FcuBektache nabylPas encore d'évaluation
- 3.09.11 NonhumanDocument6 pages3.09.11 NonhumanKìkøu KìkãmPas encore d'évaluation
- CoronavirusDocument64 pagesCoronavirusOumar m GoudienkiléPas encore d'évaluation
- 1a-Herpesviridae - Généralités - (Non Vu - )Document4 pages1a-Herpesviridae - Généralités - (Non Vu - )Ouattara BamoryPas encore d'évaluation
- hsv1 hsv2Document34 pageshsv1 hsv2HindPas encore d'évaluation
- Le DiagnosticDocument48 pagesLe DiagnosticMerya JijiPas encore d'évaluation
- Rougeole (II) - Diagnostic, TraitementDocument10 pagesRougeole (II) - Diagnostic, Traitementmahefaserge100% (1)
- PCR Directe Pour La Détection Du Génome Du Virus de La Peste Porcine Africaine À Partir de Prélèvements de Sang Sur BuvardDocument4 pagesPCR Directe Pour La Détection Du Génome Du Virus de La Peste Porcine Africaine À Partir de Prélèvements de Sang Sur BuvardCronos OttoPas encore d'évaluation
- Deficits Immunitaires-210314-Def Luc MouthonDocument41 pagesDeficits Immunitaires-210314-Def Luc MouthonLeonel TamaPas encore d'évaluation
- 3 - Diagnostic MolçculaireDocument14 pages3 - Diagnostic MolçculairePerrine ViaudPas encore d'évaluation
- PapillomavirusDocument19 pagesPapillomavirusFodié Aly CamaraPas encore d'évaluation
- RT PCRDocument15 pagesRT PCRMbackéPas encore d'évaluation
- Proteinurie Laoratoire SciencedirectDocument8 pagesProteinurie Laoratoire SciencedirectsamiPas encore d'évaluation
- Maîtriser La Dispensation Des Anticoagulants Oraux DirectsDocument4 pagesMaîtriser La Dispensation Des Anticoagulants Oraux DirectsMezouar AbdennacerPas encore d'évaluation
- Une Ordonnance Post-Infarctus Et Pontage Comportant Deux Antiagrégants PlaquéttairesDocument3 pagesUne Ordonnance Post-Infarctus Et Pontage Comportant Deux Antiagrégants PlaquéttairesMezouar AbdennacerPas encore d'évaluation
- Nouveaux Anticoagulants OrauxDocument4 pagesNouveaux Anticoagulants OrauxMezouar AbdennacerPas encore d'évaluation
- L'insuffisance Rénale Aiguë: Définition Et ÉtiologiesDocument4 pagesL'insuffisance Rénale Aiguë: Définition Et ÉtiologiesENOURLAB DrTARHLISSIA100% (1)
- Les Anticoagulants Oraux Directs Ou AODDocument10 pagesLes Anticoagulants Oraux Directs Ou AODMezouar AbdennacerPas encore d'évaluation
- L'exploration Du Rein en 2013Document15 pagesL'exploration Du Rein en 2013Mezouar AbdennacerPas encore d'évaluation
- Papillomavirus Humains (HPV) Et Cancers Associés Aspects ÉpidémiologiquesDocument8 pagesPapillomavirus Humains (HPV) Et Cancers Associés Aspects ÉpidémiologiquesMezouar AbdennacerPas encore d'évaluation
- NeurobrucelloseDocument9 pagesNeurobrucelloseMezouar AbdennacerPas encore d'évaluation
- Diagnostic Virologique D'une Hépatite Virale E (VHE)Document2 pagesDiagnostic Virologique D'une Hépatite Virale E (VHE)Mezouar AbdennacerPas encore d'évaluation
- 5'nucléotidaseDocument5 pages5'nucléotidaseMezouar AbdennacerPas encore d'évaluation
- Évaluation D'une Technique Spectrophotométrique Pour Le Dosage de L'hémoglobine Plasmatique Sur Konelab 30TMDocument8 pagesÉvaluation D'une Technique Spectrophotométrique Pour Le Dosage de L'hémoglobine Plasmatique Sur Konelab 30TMMezouar AbdennacerPas encore d'évaluation
- Manuel Technique de Microbiologie Et SérologieDocument648 pagesManuel Technique de Microbiologie Et SérologieMezouar AbdennacerPas encore d'évaluation
- Diagnostic Virologique D'une Hépatite Virale A (VHA)Document2 pagesDiagnostic Virologique D'une Hépatite Virale A (VHA)Mezouar AbdennacerPas encore d'évaluation
- Monolisa™ HBs Ag ULTRADocument14 pagesMonolisa™ HBs Ag ULTRAMezouar AbdennacerPas encore d'évaluation
- L'absorbance Comment Rendre Sa Compréhension Plus Lumineuse Aux Yeux Des Apprenant - EsDocument17 pagesL'absorbance Comment Rendre Sa Compréhension Plus Lumineuse Aux Yeux Des Apprenant - EsMezouar AbdennacerPas encore d'évaluation
- Pièges en Sérologie InfectieuseDocument4 pagesPièges en Sérologie InfectieuseMezouar AbdennacerPas encore d'évaluation
- Infections Urinaires Et Reflux Vésico-Urétéral de L'enfantDocument8 pagesInfections Urinaires Et Reflux Vésico-Urétéral de L'enfantMezouar AbdennacerPas encore d'évaluation
- ELISADocument29 pagesELISAMezouar AbdennacerPas encore d'évaluation
- Une Infection Humaine À Poxvirus L'orfDocument4 pagesUne Infection Humaine À Poxvirus L'orfMezouar AbdennacerPas encore d'évaluation
- Genscreen™ HIV-1 2 Version 2Document16 pagesGenscreen™ HIV-1 2 Version 2Mezouar AbdennacerPas encore d'évaluation
- TCF CanadaDocument7 pagesTCF CanadaAmira BenhammouPas encore d'évaluation
- V120i 25324 5 PM FrenchDocument450 pagesV120i 25324 5 PM FrenchFodilsbsPas encore d'évaluation
- TD 3 Capteur Et Instrumentation 2021 CorrigéDocument4 pagesTD 3 Capteur Et Instrumentation 2021 CorrigéRabah Hadjar100% (5)
- Texte 2Document7 pagesTexte 2Ashema Tshokama100% (1)
- 1 13 Imprimer Documents RecapitulatifsDocument14 pages1 13 Imprimer Documents RecapitulatifsAzsrtyPas encore d'évaluation
- DAF Comite Audit CAadmin IFA DFCGDocument8 pagesDAF Comite Audit CAadmin IFA DFCGToah Bi Marc DidiaPas encore d'évaluation
- Lexium SD3 - LU9GC3Document2 pagesLexium SD3 - LU9GC3Mohamed Amine LABIDIPas encore d'évaluation
- Synthèse CinématiqDocument1 pageSynthèse CinématiqmasteratsiiPas encore d'évaluation
- 2022 DEPREZ-AudreyDocument38 pages2022 DEPREZ-Audreywb5510078Pas encore d'évaluation
- Langage Et AnthropologieDocument20 pagesLangage Et AnthropologieEl Marjani MokhtarPas encore d'évaluation
- Cours1 MasterDocument13 pagesCours1 Mastermahfoud1254Pas encore d'évaluation
- Fe 50 Fiche Explicative Des Fiches Cee Sme v17 VFDocument19 pagesFe 50 Fiche Explicative Des Fiches Cee Sme v17 VFhassankchPas encore d'évaluation
- Cours #7 - La Loupe - 10 - 01Document11 pagesCours #7 - La Loupe - 10 - 01Said MessaoudPas encore d'évaluation
- (Aritech) Cd3402s3Plus PDFDocument83 pages(Aritech) Cd3402s3Plus PDFhenryPas encore d'évaluation
- Adaptation de La Tension À L'utilisationDocument7 pagesAdaptation de La Tension À L'utilisationfatima zohraPas encore d'évaluation
- AccumulateurDocument28 pagesAccumulateurdarcisPas encore d'évaluation
- OranunevillealgriennereconquiseuncentrehistoriqueenmutationDocument16 pagesOranunevillealgriennereconquiseuncentrehistoriqueenmutationsidoPas encore d'évaluation
- Banque CCP Sup ProbaDocument2 pagesBanque CCP Sup ProbaAziz ChafikPas encore d'évaluation
- Serie2TLAIng2013 2014Document2 pagesSerie2TLAIng2013 2014Ghazouani HaythemPas encore d'évaluation
- Soupe Choux Fleur CheddarDocument3 pagesSoupe Choux Fleur CheddarLoren ZoPas encore d'évaluation
- Guide Du Projet Professionnel Et Personnel de L'étudiantDocument32 pagesGuide Du Projet Professionnel Et Personnel de L'étudiantTik Tok Galsen Fun OfficielPas encore d'évaluation
- Pfe GC 0047 PDFDocument105 pagesPfe GC 0047 PDFHervé JabeaPas encore d'évaluation